はじめに
楽なので、Rのplot()時に、レインボー色(grDevices::rainbow)を使うんだけど、 お世辞にも良い色とは言えず、ドギツイ色を使うねと周囲から言われる。
今回、それを挽回するために、そのレインボー色を和らげる方法を紹介する。
rainbow関数では、16進数で表される「カラーコード」*1 + 透明度が出力される。 そのため、透明度の設定を変更すると、程良いレインボー・カラーにすることができる。
例えば、grDevices::rainbow(10)の出力結果は、次のようになる。
grDevices::rainbow(10) [1] "#FF0000FF" "#FF9900FF" "#CCFF00FF" [4] "#33FF00FF" "#00FF66FF" "#00FFFFFF" [7] "#0066FFFF" "#3300FFFF" "#CC00FFFF" [10] "#FF0099FF"
最後の2桁 FF は、透明度ゼロ(非透明)という意味になるので、
そこを数字に置き換える。
この文字列処理には、文字列の部分抽出ができる、base::substr関数を使用する。 substr関数で、カラーコードの1文字目から7文字目までを抜き出して、 任意の数字2桁とくっ付ければ、色変換ができる。
実際に、FF、80、60、40、20でのプロット結果を見てみる*2。
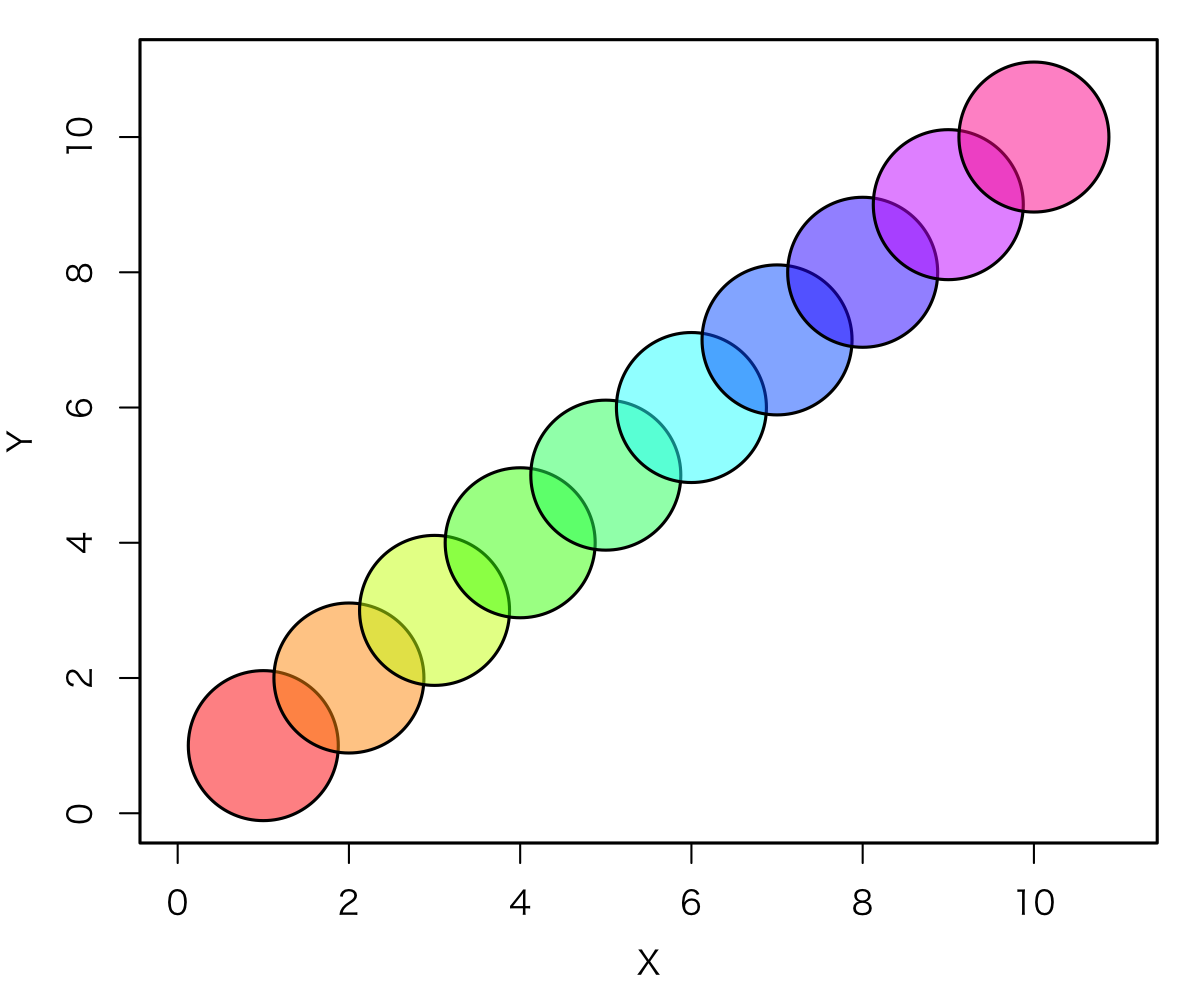
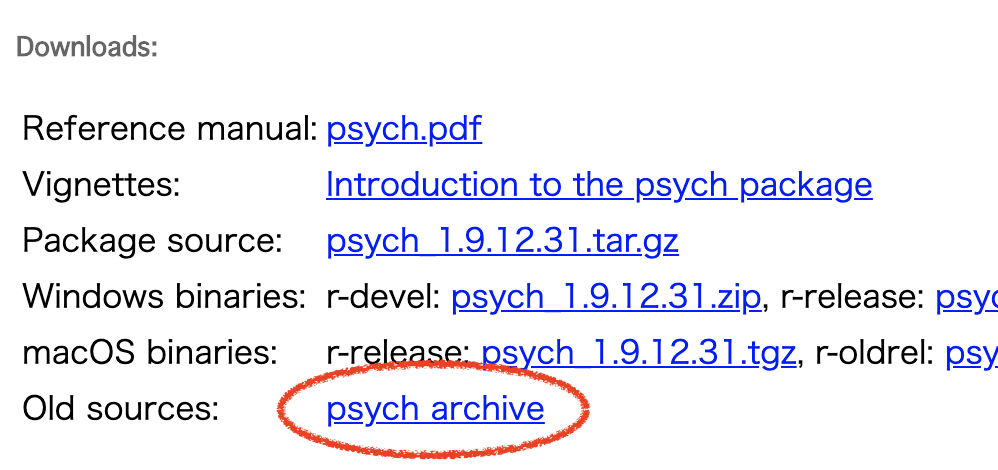
レインボー色(grDevices::rainbow関数)の設定
レインボー・カラー 透明度 FF
#プロットデータ Data <- data.frame(X=1:10, Y=1:10) #par設定 par(family="HiraKakuProN-W3", lwd=1.5, xpd=F, mgp=c(2.5, 1, 0), mai=c(0.7,0.7, 0.2, 0.2)) #レインボー・カラー 透明度 FF col100 <- rainbow(10) col100 #上記と同じ # [1] "#FF0000FF" "#FF9900FF" "#CCFF00FF" # [4] "#33FF00FF" "#00FF66FF" "#00FFFFFF" # [7] "#0066FFFF" "#3300FFFF" "#CC00FFFF" #[10] "#FF0099FF" plot(Data, cex=10, pch=21, col=1, bg=col100, xlim=c(0,11), ylim=c(0,11)) #図保存 for Mac #quartz.save(paste("C100.png", sep=""), type = "png", dpi = 200)
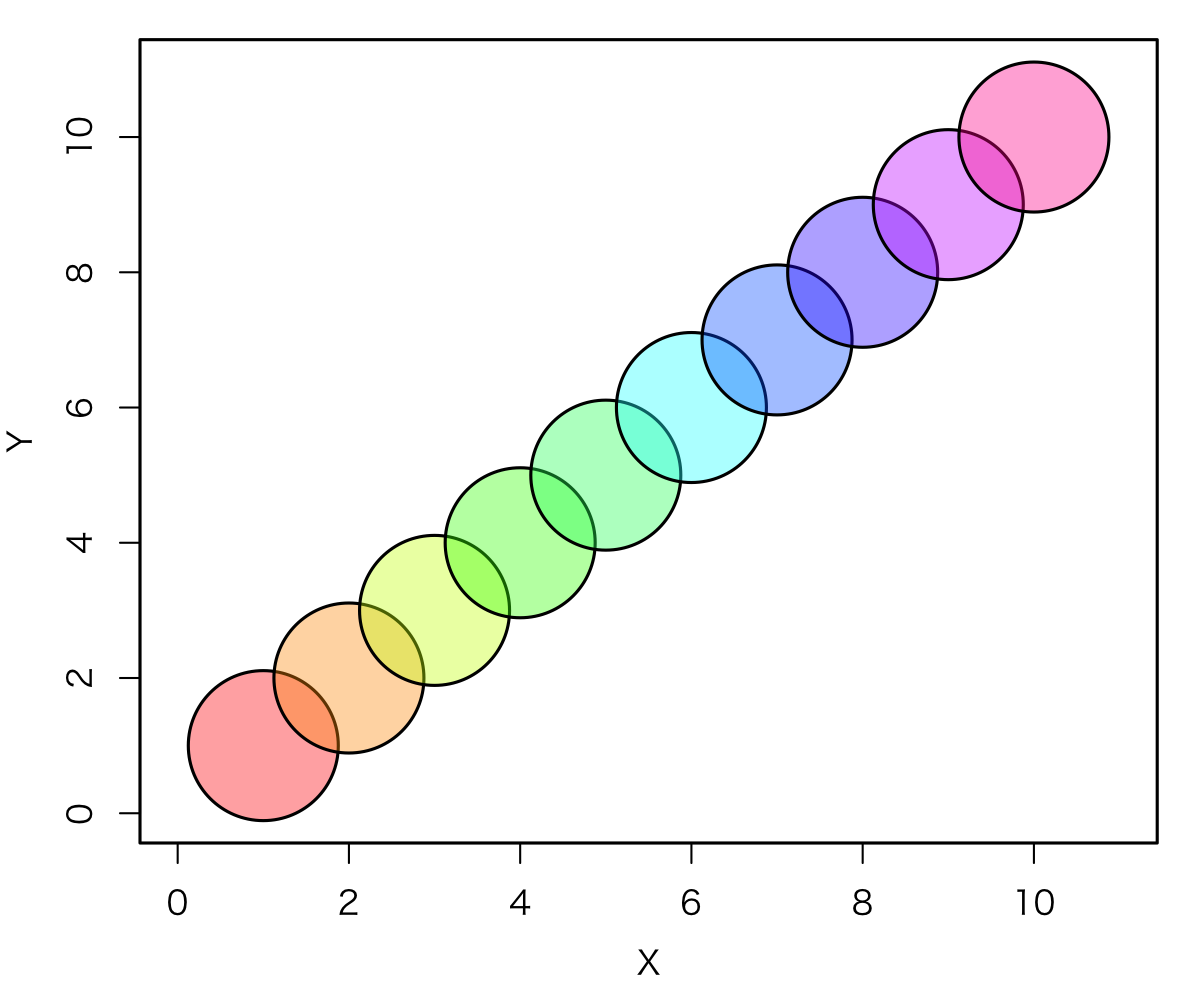
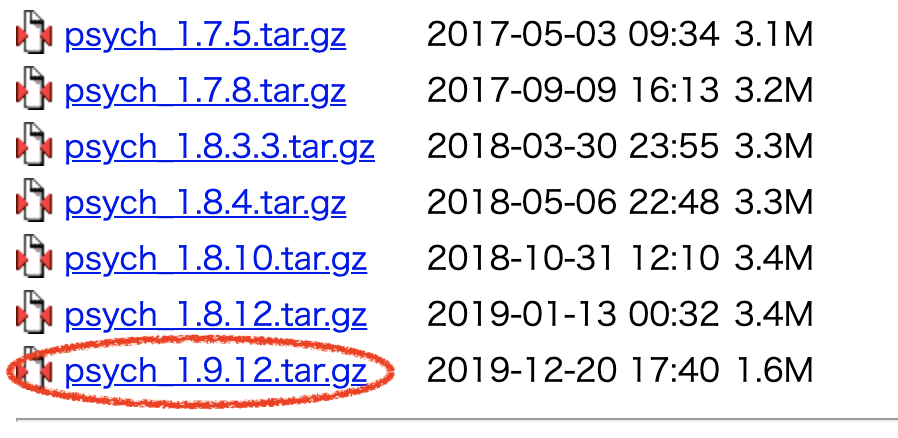
レインボー・カラー 透明度 80
#レインボー・カラー 透明度 80 col80 <- paste(substr(rainbow(10), 1, 7), "80", sep="") col80 # [1] "#FF000080" "#FF990080" "#CCFF0080" # [4] "#33FF0080" "#00FF6680" "#00FFFF80" # [7] "#0066FF80" "#3300FF80" "#CC00FF80" #[10] "#FF009980" plot(Data, cex=10, pch=21, col=1, bg=col80, xlim=c(0,11), ylim=c(0,11)) #quartz.save(paste("C080.png", sep=""), type = "png", dpi = 200)
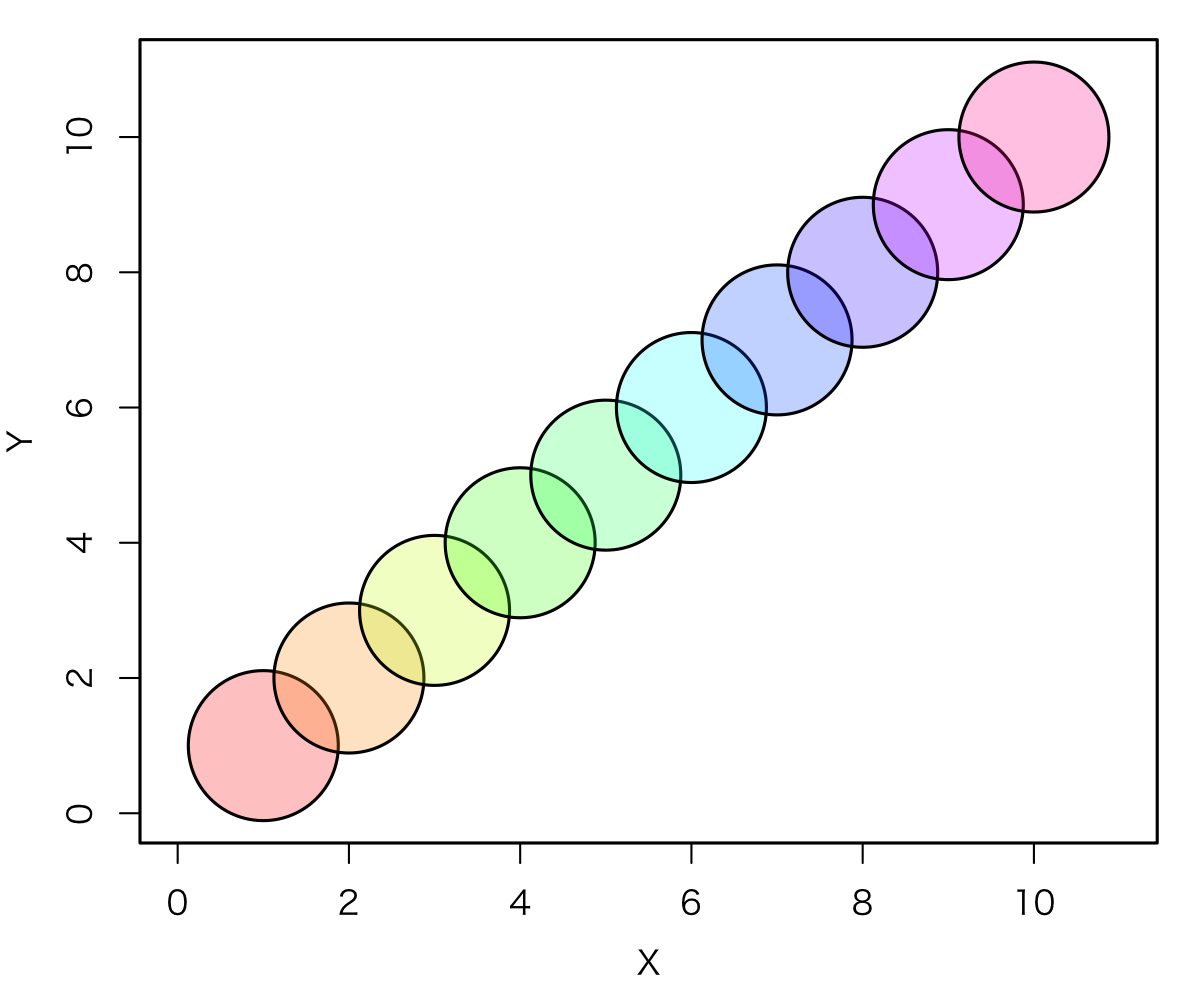
レインボー・カラー 透明度 60
#レインボー・カラー 透明度 60 col60 <- paste(substr(rainbow(10), 1, 7), "60", sep="") col60 # [1] "#FF000060" "#FF990060" "#CCFF0060" # [4] "#33FF0060" "#00FF6660" "#00FFFF60" # [7] "#0066FF60" "#3300FF60" "#CC00FF60" #[10] "#FF009960" plot(Data, cex=10, pch=21, col=1, bg=col60, xlim=c(0,11), ylim=c(0,11)) #quartz.save(paste("C060.png", sep=""), type = "png", dpi = 200)
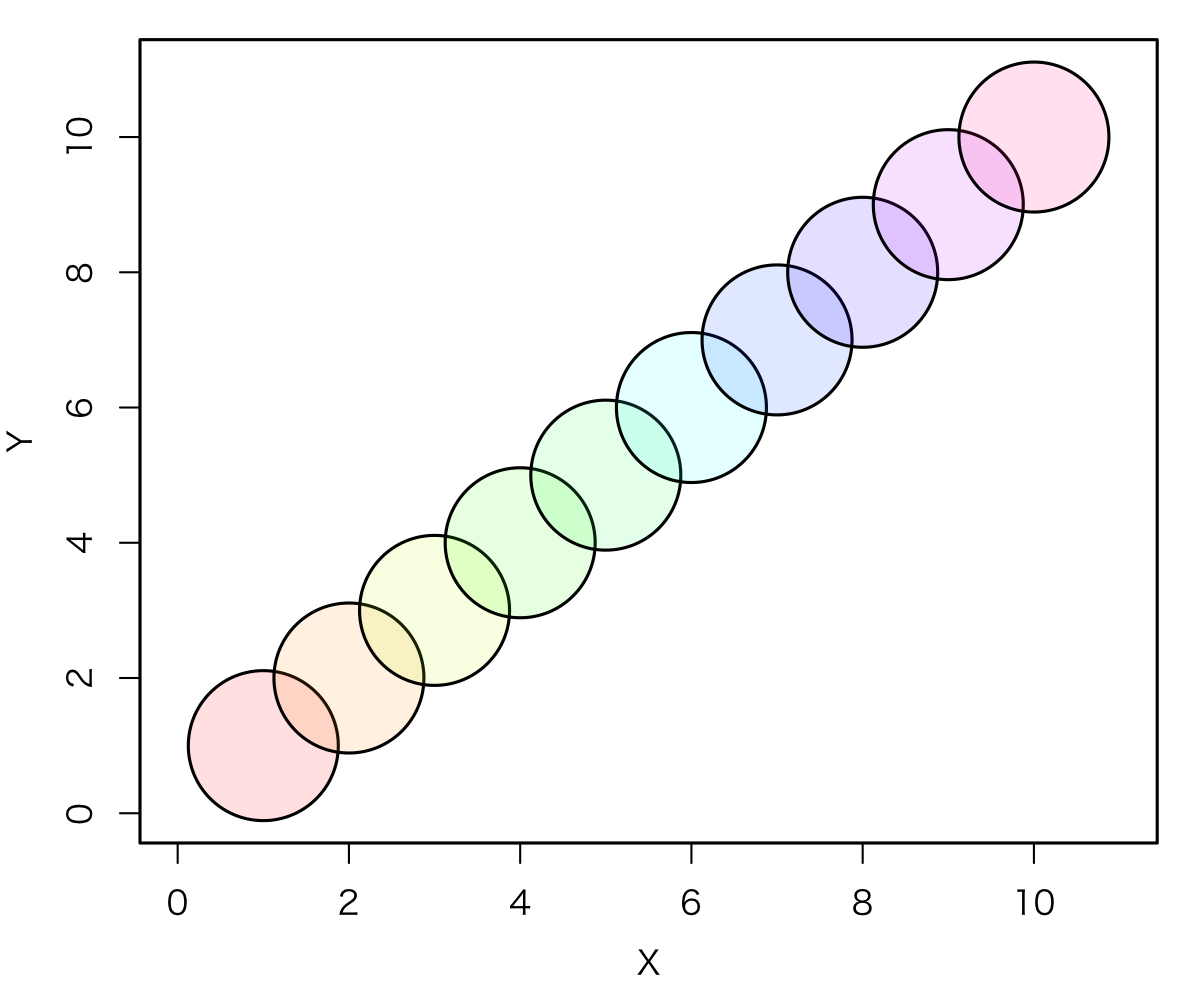
レインボー・カラー 透明度 40
#レインボー・カラー 透明度 40 col40 <- paste(substr(rainbow(10), 1, 7), "40", sep="") col40 # [1] "#FF000040" "#FF990040" "#CCFF0040" # [4] "#33FF0040" "#00FF6640" "#00FFFF40" # [7] "#0066FF40" "#3300FF40" "#CC00FF40" #[10] "#FF009940" plot(Data, cex=10, pch=21, col=1, bg=col40, xlim=c(0,11), ylim=c(0,11)) #quartz.save(paste("C040.png", sep=""), type = "png", dpi = 200)
レインボー・カラー 透明度 20
#レインボー・カラー 透明度 20 col20 <- paste(substr(rainbow(10), 1, 7), "20", sep="") col20 # [1] "#FF000020" "#FF990020" "#CCFF0020" # [4] "#33FF0020" "#00FF6620" "#00FFFF20" # [7] "#0066FF20" "#3300FF20" "#CC00FF20" #[10] "#FF009920" plot(Data, cex=10, pch=21, col=1, bg=col20, xlim=c(0,11), ylim=c(0,11)) #quartz.save(paste("C020.png", sep=""), type = "png", dpi = 200)
Rで使える他のカラーパレットについて
palette()関数を使います。
R4.0以降、デフォルトのカラーパレットが変わっています。 新しいパレットでは以前同様の色相を使用していますが、 輝度の点でよりバランスが取れており、極端に派手な色合いを避けています。
palette()関数のデフォルトカラー 透明度 FF
#プロットデータ Data <- data.frame(X=1:8, Y=1:8) #par設定 par(family="HiraKakuProN-W3", lwd=1.5, xpd=F, mgp=c(2.5, 1, 0), mai=c(0.7,0.7, 0.2, 0.2)) #デフォルト・パレット pal100 <- palette() pal100 #[1] "black" "#DF536B" "#61D04F" "#2297E6" "#28E2E5" #[6] "#CD0BBC" "#F5C710" "gray62" plot(Data, cex=10, pch=21, col=1, bg=pal100, xlim=c(0,11), ylim=c(0,11)) #図保存 for Mac #quartz.save(paste("P100.png", sep=""), type = "png", dpi = 200)

palette()関数のデフォルトカラー 透明度 75
#プロットデータ Data <- data.frame(X=1:8, Y=1:8) #par設定 par(family="HiraKakuProN-W3", lwd=1.5, xpd=F, mgp=c(2.5, 1, 0), mai=c(0.7,0.7, 0.2, 0.2)) #デフォルト・パレット 75 pal75 <- paste(c("#000000", palette()[c(-1, -8)], "#9e9e9e"), "75", sep="") pal75 #[1] "#00000075" "#F8766D75" "#00BA3875" "#619CFF75" #[5] "#00BFC475" "#F564E375" "#B79F0075" "#9e9e9e75" plot(Data, cex=10, pch=21, col=1, bg=pal75, xlim=c(0,11), ylim=c(0,11)) #図保存 for Mac #quartz.save(paste("P75.png", sep=""), type = "png", dpi = 200)

まとめ
プロットの種類によるけど、透明度は60くらいが良さそうに思う。
よって、paste(substr(rainbow(10), 1, 7), "60", sep="")を推奨。
補足
実は、もっと簡単には、rainbowの引数であるalpha値を設定しよう。
alpha値を「1より小さい値」に設定することで、半透明の設定ができる。
以下が実施例である。
alpha値 0.5 で、半透明の設定
#データの準備 Data <- data.frame(X=1:10, Y=1:10) #alpha値 0.5 で、半透明の設定 col50 <- rainbow(10, alpha =0.5) par(family="HiraKakuProN-W3", lwd=1.5, xpd=T, mgp=c(2.5, 1, 0), mai=c(0.7,0.7, 0.2, 0.2)) plot(Data, cex=10, pch=21, col=1, bg=col50, xlim=c(0,11), ylim=c(0,11)) quartz.save(paste("C050.png", sep=""), type = "png", dpi = 200)

レインボーを逆順にする
#レインボーを逆順にする col50_r <- rainbow(10, alpha =0.5, rev = T) par(family="HiraKakuProN-W3", lwd=1.5, xpd=T, mgp=c(2.5, 1, 0), mai=c(0.7,0.7, 0.2, 0.2)) plot(Data, cex=10, pch=21, col=1, bg=col50_r, xlim=c(0,11), ylim=c(0,11)) quartz.save(paste("C050_r.png", sep=""), type = "png", dpi = 200)

ちょっと落ち着いた感じのレインボー・プロット
#ちょっと落ち着いた感じのレインボー・プロット library(colorspace) ColorSpa <- colorspace::rainbow_hcl(10, alpha = 0.5) par(family="HiraKakuProN-W3", lwd=1.5, xpd=T, mgp=c(2.5, 1, 0), mai=c(0.7,0.7, 0.2, 0.2)) plot(Data, cex=10, pch=21, col=1, bg=ColorSpa, xlim=c(0,11), ylim=c(0,11)) quartz.save(paste("C050_ColorSpa.png", sep=""), type = "png", dpi = 200)

【Rのジミ〜な小技シリーズ】
*1:https://woma2.com/design/colorcodes-post-1203/
*2:透明度が分かるよう、あえてマーカーを重ねてみた